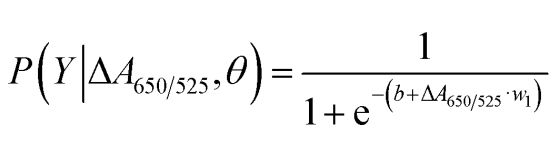La detección temprana del cáncer mediante biopsia líquida es una nueva línea de investigación en la detección y el diagnóstico del cáncer, propuesta por el Instituto Nacional del Cáncer de EE. UU. en los últimos años, con el objetivo de detectar cáncer en etapa temprana o incluso lesiones precancerosas. Se ha utilizado ampliamente como un nuevo biomarcador para el diagnóstico temprano de diversas neoplasias malignas, como cáncer de pulmón, tumores gastrointestinales, gliomas y tumores ginecológicos.
La aparición de plataformas para identificar biomarcadores del panorama de metilación (Methylscape) tiene el potencial de mejorar significativamente la detección temprana del cáncer existente, colocando a los pacientes en la etapa tratable más temprana.
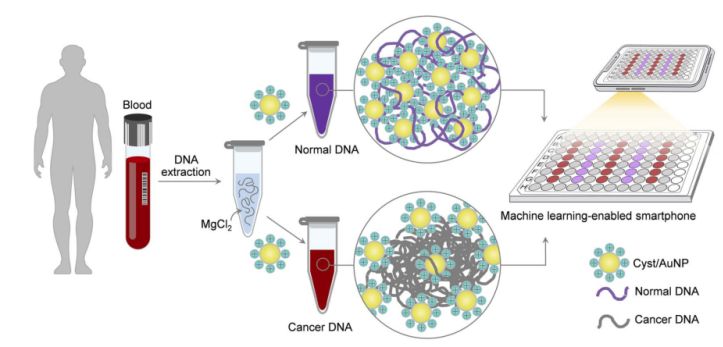
Recientemente, investigadores han desarrollado una plataforma de detección sencilla y directa para la detección del paisaje de metilación, basada en nanopartículas de oro con recubrimiento de cisteamina (Cyst/AuNP), combinada con un biosensor para smartphones. Esta plataforma permite la detección temprana y rápida de una amplia gama de tumores. La detección temprana de leucemia puede realizarse en los 15 minutos siguientes a la extracción de ADN de una muestra de sangre, con una precisión del 90 %. El título del artículo es: Detección rápida de ADN canceroso en sangre humana mediante AuNP con recubrimiento de cisteamina y un smartphone con aprendizaje automático.
Figura 1. Una plataforma de detección simple y rápida para la detección del cáncer a través de componentes Cyst/AuNPs se puede lograr en dos simples pasos.
Esto se muestra en la Figura 1. Primero, se utilizó una solución acuosa para disolver los fragmentos de ADN. Posteriormente, se añadieron quistes/AuNPs a la solución mixta. El ADN normal y el maligno presentan diferentes propiedades de metilación, lo que resulta en fragmentos de ADN con distintos patrones de autoensamblaje. El ADN normal se agrega de forma laxa y, finalmente, agrupa quistes/AuNPs, lo que da lugar a la naturaleza desplazada al rojo de los quistes/AuNPs, de modo que se puede observar a simple vista un cambio de color de rojo a púrpura. Por el contrario, el perfil de metilación único del ADN canceroso conduce a la producción de grupos más grandes de fragmentos de ADN.
Se tomaron imágenes de placas de 96 pocillos con la cámara de un teléfono inteligente. El ADN del cáncer se midió con un teléfono inteligente equipado con aprendizaje automático, comparándolo con métodos espectroscópicos.
Detección del cáncer en muestras de sangre reales
Para ampliar la utilidad de la plataforma de detección, los investigadores aplicaron un sensor que distinguió con éxito entre ADN normal y canceroso en muestras de sangre real. Los patrones de metilación en los sitios CpG regulan epigenéticamente la expresión génica. En casi todos los tipos de cáncer, se ha observado una alternancia de cambios en la metilación del ADN y, por lo tanto, en la expresión de genes que promueven la tumorigénesis.
Como modelo para otros cánceres asociados con la metilación del ADN, los investigadores utilizaron muestras de sangre de pacientes con leucemia y controles sanos para investigar la eficacia del panorama de metilación en la diferenciación de cánceres leucémicos. Este biomarcador del panorama de metilación no solo supera a los métodos actuales de cribado rápido de leucemia, sino que también demuestra la viabilidad de extenderlo a la detección temprana de una amplia gama de cánceres mediante este ensayo simple y directo.
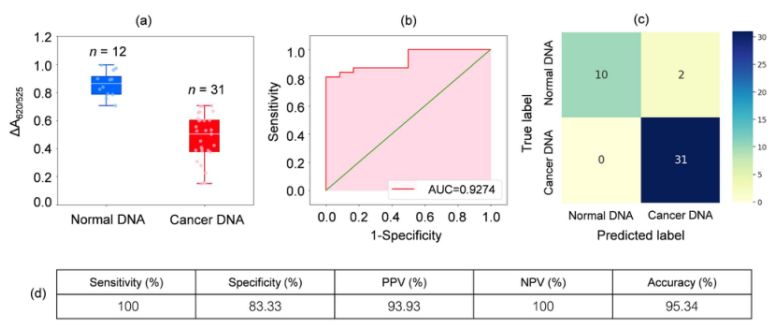
Se analizó el ADN de muestras de sangre de 31 pacientes con leucemia y 12 individuos sanos. Como se muestra en el diagrama de cajas de la Figura 2a, la absorbancia relativa de las muestras de cáncer (ΔA650/525) fue menor que la del ADN de muestras normales. Esto se debió principalmente a la mayor hidrofobicidad, que condujo a una densa agregación del ADN canceroso, lo que impidió la agregación de quistes/AuNP. Como resultado, estas nanopartículas se dispersaron completamente en las capas externas de los agregados de cáncer, lo que resultó en una dispersión diferente de los quistes/AuNP adsorbidos en los agregados de ADN normal y canceroso. Se generaron curvas ROC variando el umbral desde un valor mínimo de ΔA650/525 hasta un valor máximo.
Figura 2.(a) Valores de absorbancia relativa de soluciones de quistes/AuNPs que muestran la presencia de ADN normal (azul) y canceroso (rojo) en condiciones optimizadas.
(DA650/525) de diagramas de caja; (b) Análisis ROC y evaluación de pruebas diagnósticas. (c) Matriz de confusión para el diagnóstico de pacientes normales y con cáncer. (d) Sensibilidad, especificidad, valor predictivo positivo (VPP), valor predictivo negativo (VPN) y precisión del método desarrollado.
Como se muestra en la Figura 2b, el área bajo la curva ROC (AUC = 0,9274) obtenida para el sensor desarrollado mostró alta sensibilidad y especificidad. Como se puede observar en el diagrama de cajas, el punto más bajo, que representa el grupo de ADN normal, no está bien separado del punto más alto, que representa el grupo de ADN canceroso; por lo tanto, se utilizó una regresión logística para diferenciar entre los grupos normal y canceroso. Dado un conjunto de variables independientes, se estima la probabilidad de que ocurra un evento, como un grupo de cáncer o normal. La variable dependiente oscila entre 0 y 1. Por lo tanto, el resultado es una probabilidad. Determinamos la probabilidad de identificación de cáncer (P) con base en ΔA650/525 de la siguiente manera.
donde b = 5,3533, w1 = -6,965. Para la clasificación de la muestra, una probabilidad inferior a 0,5 indica una muestra normal, mientras que una probabilidad de 0,5 o superior indica una muestra con cáncer. La Figura 2c muestra la matriz de confusión generada a partir de la validación cruzada sin modificaciones, utilizada para validar la estabilidad del método de clasificación. La Figura 2d resume la evaluación diagnóstica del método, incluyendo la sensibilidad, la especificidad, el valor predictivo positivo (VPP) y el valor predictivo negativo (VPN).
Biosensores basados en teléfonos inteligentes
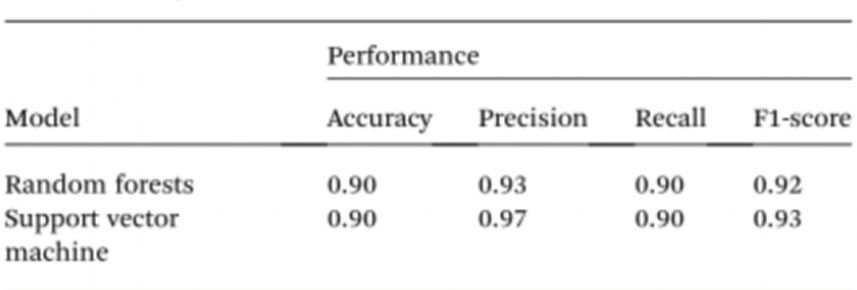
Para simplificar aún más las pruebas de muestras sin el uso de espectrofotómetros, los investigadores utilizaron inteligencia artificial (IA) para interpretar el color de la solución y distinguir entre individuos normales y cancerosos. Dado esto, se utilizó visión artificial para traducir el color de la solución de quistes/AuNPs en ADN normal (púrpura) o ADN canceroso (rojo) utilizando imágenes de placas de 96 pocillos tomadas a través de una cámara de teléfono móvil. La inteligencia artificial puede reducir los costos y mejorar la accesibilidad en la interpretación del color de las soluciones de nanopartículas, y sin el uso de ningún accesorio de hardware óptico para teléfonos inteligentes. Finalmente, se entrenaron dos modelos de aprendizaje automático, incluyendo Random Forest (RF) y Support Vector Machine (SVM) para construir los modelos. Tanto los modelos RF como SVM clasificaron correctamente las muestras como positivas y negativas con una precisión del 90,0%. Esto sugiere que el uso de inteligencia artificial en la biodetección basada en teléfonos móviles es bastante posible.
Figura 3.(a) Clase objetivo de la solución registrada durante la preparación de la muestra para el paso de adquisición de imágenes. (b) Imagen de ejemplo tomada durante el paso de adquisición de imágenes. (c) Intensidad de color de la solución de quiste/AuNPs en cada pocillo de la placa de 96 pocillos extraída de la imagen (b).
Utilizando Cyst/AuNPs, investigadores han desarrollado con éxito una plataforma de detección sencilla para la detección del paisaje de metilación y un sensor capaz de distinguir el ADN normal del ADN canceroso al utilizar muestras de sangre real para el cribado de leucemia. El sensor desarrollado demostró que el ADN extraído de muestras de sangre real era capaz de detectar de forma rápida y rentable pequeñas cantidades de ADN canceroso (3 nM) en pacientes con leucemia en 15 minutos, con una precisión del 95,3 %. Para simplificar aún más el análisis de muestras eliminando la necesidad de un espectrofotómetro, se utilizó aprendizaje automático para interpretar el color de la solución y diferenciar entre individuos normales y cancerosos mediante una fotografía tomada con un teléfono móvil, con una precisión del 90 %.
Referencia: DOI: 10.1039/d2ra05725e
Hora de publicación: 18 de febrero de 2023
 中文网站
中文网站